商家描述
售后服务
产品评价(0)
一、介绍
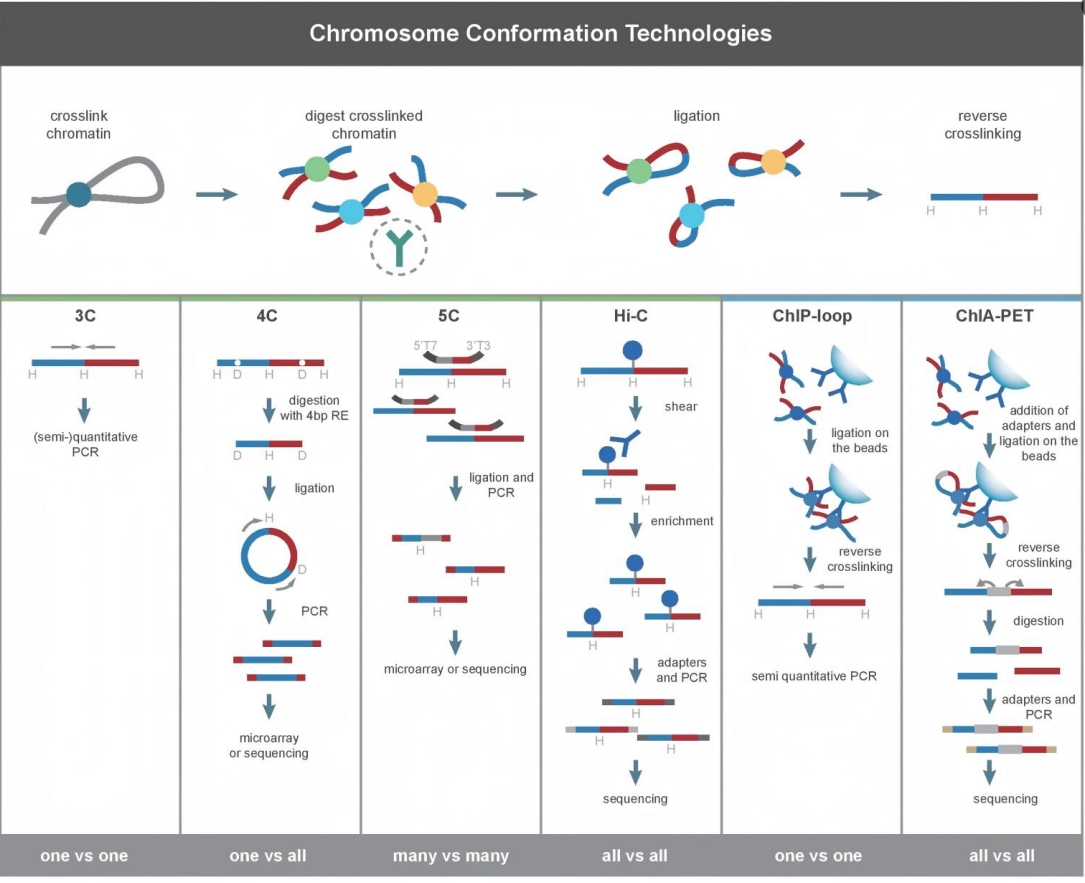
Hi-C 技术源于基因组捕获技术(Chromosome conformation capture,3C),用于分析染色质三维空间结构的一种测序方法。
用途:
- 量化在三维空间中基因组的染色质间交联(cross-linked chromatin)
- 解析全基因组互作模式,如启动子和增强子互作
- 构建三维空间结构模型,如研究基因组三维结构特征:compartment,TAD,loop等
- 构建全基因组互作图谱
- 辅助提升基因组组装
- 构建基因组单体型图谱
二、原理及步骤
1、甲醛固定
先加入甲醛将基因组中参与染色质互作作用的蛋白质凝固
一般将活体样本在室温用 1-3%的甲醛处理 10-30min,但是此步骤会减少限制内切酶对DNA序列的消化效率,需要严格控制。
2、酶切序列
用限制性内切酶切割基因组,打断后的片段大小会影响测序分辨率,一般有两种酶可供选择:6bp 的限制性内切酶,4bp 的限制性内切酶。后者具有更高的分辨率。EcoR1 或 HindIII 用于每4000bp切割一次基因组,在人类基因组中产生约100万个片段。
3、末端修复
得到的片段具有平末端或粘性末端,然后将末端补平修复,加入生物素。
4、连接及解交联
使用 T4 DNA连接酶连接互作片段,形成环状。将连接DNA片段的蛋白质消化掉,得到交联片段。
5、序列打断
使用超声波或其他方式,再次打断片段。
6、上机测序
用磁珠将带生物素的捕获,制作文库,上机测序。
 会员登录
会员登录.getTime()%>)
 购物车()
购物车()

 成功收藏产品
成功收藏产品