产品概述
产品描述
SpCas9是一种由crRNA和tracrRNA共同(或两者融合后形成的sgRNA)引导的DNA内切核酸酶,来源于S. pyogenes菌株。在靶标双链DNA存在PAM(5′-NGG-3′)的情况下,特异地剪切靶标双链DNA,使DNA双链断裂并生成平末端。PAM对于Cas9的识别和剪切都是必需的,其剪切位点位于目标序列(target sequence)内,离PAM区3个碱基。SpCas9酶的HNH和RuvC结构域分别负责切割靶标双链DNA中与sgRNA配对和非配对的链。本产品中,SpCas9的H840和D10都被突变为A,从而使其HNH和RuvC功能域失活,获得了Sp-dCas9酶。因此,Sp-dCas9只有结合靶标DNA的活性,而没有切割靶标DNA的活性。
产品组分
|
组分 |
32102-01(100 pmol) |
32102-03(1,000 pmol) |
|
|
100 pmol | 1,000 pmol |
|
|
1 mL | 1 mL |
a.Sp-dCas9 Nuclease浓度为10 μM,即10 pmol/μL,100 pmol规格的总体积为10 μL,1,000 pmol规格的总体积为100 μL;
保存条件
-20℃储存;≤ 0℃运输。
产品优势
✽ 高纯度
✽ 高灵敏度
✽ 高特异性
实验案例
Sp-dCas9与靶标DNA的结合实验:为了验证Sp-dCas9与target DNA的结合,我们通过5’端带FAM基团的引物在全长59 bp的target dsDNA扩增产物末端引入FAM标记。然后将target dsDNA-FAM与对应的sgRNA、Sp-dCas9 Nuclease加入结合反应体系,在37℃静置孵育30 min。最后,用2%的琼脂糖凝胶低温电泳(100 V,60 min),在化学发光成像分析仪(GE ImageQuantLAS4000)上用荧光通道检测样本条带。实验结果表明,Sp-dCas9 + sgRNA + target dsDNA-FAM泳道电泳跑胶迁移速率较慢,说明Sp-dCas9在sgRNA的引导下特异地识别并结合了target dsDNA。此外,还可以用非变性聚丙烯酰胺凝胶电泳(Native-PAGE)对Sp-dCas9与target DNA的结合进行检测。
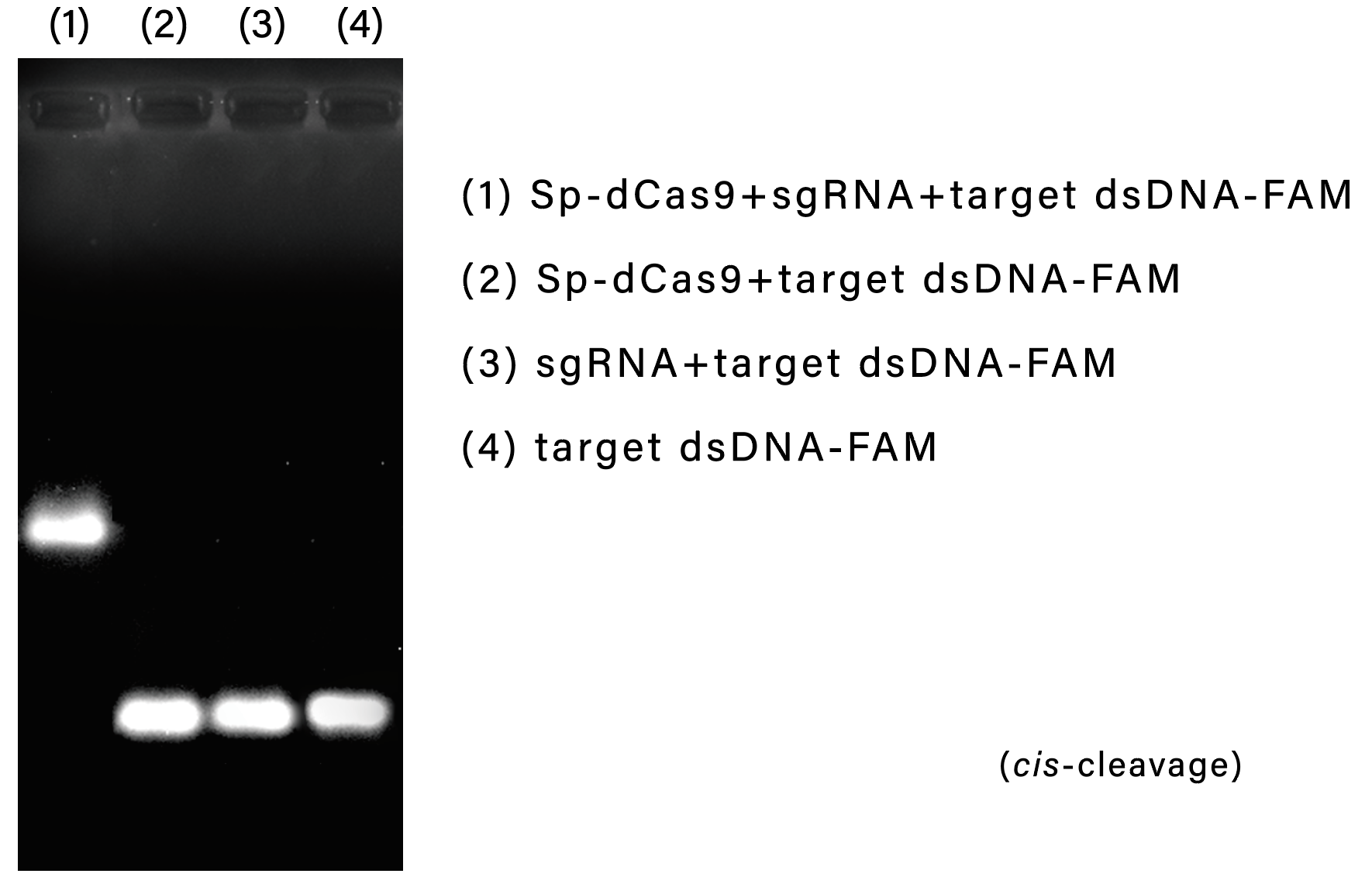
图1:Sp-dCas9在sgRNA的引导下特异地识别并结合target dsDNA
实验流程
1. Sp-dCas9与靶标DNA结合实验
|
组分 |
体积 |
终浓度 |
|
10 × HOLMES Buffer for Cas9 |
2 μL | 1 × |
|
10 μM Sp-dCas9 Nuclease |
1.5 μL | 750 nM |
|
10 μM sgRNA |
1.5 μL | 750 nM |
|
1 μM Target dsDNA |
3 μL | 150 nM |
|
Nuclease-free water |
Up to 20 μL |
◆ 37℃反应30 min,可通过非变性核酸电泳对实验结果进行检测。
常见问题解答
Q1: dCas9与Cas9有什么区别?
A1: Cas9可识别并剪切dsDNA靶标,其机制涉及HNH和RuvC两个结构域,HNH剪切TS链,RuvC剪切NTS链,两者共同产生dsDNA双链断裂(DSBs),产生平末端。但dCas9由于H840A和D10A氨基酸突变,导致HNH和RuvC结构域失活,只能结合靶标dsDNA,而不能切割靶标dsDNA。此外,还有nCas9,由于D10A突变导致RuvC结构域失活,nCas9结合dsDNA后只能剪切其中的一条链形成切口。
Q2: Sp-dCas9的sgRNA应该如何设计?
A2: Sp-dCas9的sgRNA可参照以下序列设计:
5′–NNNNNNNNNNNNNNNNNNNNGUUUUAGAGCUAUGCUGAAAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUG–3′,(下划线表示与特异靶标序列互补配对的spacer区)。
 会员登录
会员登录.getTime()%>)
 购物车()
购物车()


 成功收藏产品
成功收藏产品
 Sp-dCas9 Nuclease (10 μM)
Sp-dCas9 Nuclease (10 μM)  10 × HOLMES Buffer for Cas9
10 × HOLMES Buffer for Cas9